检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
检测到您当前使用浏览器版本过于老旧,会导致无法正常浏览网站;请您使用电脑里的其他浏览器如:360、QQ、搜狗浏览器的极速模式浏览,或者使用谷歌、火狐等浏览器。
 下载Firefox
下载Firefox
线粒体是细胞中的“动力工厂”,与核基因组不同,线粒体基因组较小且呈环状,仅包含少量基因,然而其突变却可能导致一系列遗传性疾病1。线粒体DNA与核基因组DNA由线粒体膜和核膜分别包裹,互相隔绝。然而在“内共生学说”中,原核生物来源的线粒体基因组DNA随着生物演化逐渐转移到了核基因组DNA中2。这些核基因组上的线粒体DNA片段(nuclear-mitochondrial DNA segments, NUMTs),近年来在人类基因组中也被广泛发现。不仅如此,此前的研究发现大约104的人群中就会发生1个新的NUMT,而在癌症样品中新的NUMT发生频率大幅升高3。近年来,基因编辑领域从核基因组编辑逐渐拓展到线粒体DNA编辑。随着线粒体定向转录激活因子核酸酶(mitoTALEN)和新型碱基编辑器DdCBE的开发,特异清除带有致病性突变的线粒体或修正线粒体基因突变成为了可能4。然而在核基因组靶向编辑的过程中,由于DNA双链断裂(DNA double-stranded breaks, DSBs)的发生,靶向位点可能发生大片段缺失、插入、染色体易位等异常情况引发基因组的不稳定。在这一前提下,线粒体DNA的稳定性在基因编辑过程中是否会受到影响,仍是一个未解之谜。
2024年11月1日,北京大学生命科学学院和北大-清华生命科学联合中心胡家志课题组及合作者在Nature Communications发表了标题为:Transfer of mitochondrial DNA into the nuclear genome during induced DNA breaks的研究论文。该项研究中作者采用实验室前期开发的用于系统分析基因编辑产物的高通量测序方法——PEM-seq5(Cell Discovery | 胡家志研究组开发出优化基因编辑和追踪DNA修复的新方法),发现在核基因组编辑过程中,线粒体DNA片段可能插入到靶向位点,与此同时线粒体DNA的靶向编辑也会引发线粒体DNA的不稳定,导致其插入到核基因组中。作者还发现,通过同时表达DNA外切酶TREX1或TREX2可以降低线粒体DNA插入的频率。这一研究通过研究基因编辑引发的DNA断裂对线粒体DNA的影响,揭示了基因编辑过程中线粒体DNA片段插入核基因组带来的潜在风险并提出了可行的解决思路。
作者研究利用PEM-seq方法分析了CRISPR-Cas编辑核基因组后靶向位点的情况,发现在多种CRISPR-Cas编辑酶及Cas9家族变体编辑后,核基因组编辑位点都发生了mtDNA与靶向位点融合的序列(图一A,B)。在编辑后的细胞中mtDNA与核基因组靶向位点融合的事件在每103至105次编辑事件中发生一次,而未编辑的样本几乎没有mt-nuclear DNA融合,说明这些事件主要与CRISPR-Cas编辑相关。与SpCas9相比,Cas12家族的编辑工具表现出稍低的mtDNA整合水平,而使用碱基编辑器可以通过降低DSB频率大幅降低mtDNA整合的频率。不仅如此,作者在编辑过后的CAR T细胞体系,小鼠TCR T细胞,以及碱基编辑后的小鼠胚胎中都发现了由基因编辑引起的线粒体DNA与核基因组融合事件。
图一. PEM-seq检测线粒体DNA片段在核基因组靶向位点的插入
作者进一步通过特异性扩增靶向位点的插入序列证明在基因编辑过程中线粒体DNA碎片可以插入到核基因组的靶向位点,且线粒体药物CCCP和Paraquat处理或在线粒体引入DSB都能显著增加核基因组靶向位点mtDNA的插入,说明这一事件频率受到线粒体DNA稳定性的影响(图一C,D)。
那么由于线粒体基因编辑导致的线粒体DNA碎片是否有可能插入到核基因组中呢?作者利用PEM-seq发现,在mitoTALEN编辑后线粒体DNA与核基因组整合序列的断点大多发生在mitoTALEN的编辑窗口内。同样的,在DdCBE编辑后也能发现编辑位点与核基因组的整合,证明线粒体编辑器同样可以导致线粒体DNA插入到核基因组中(图二A)。在细胞中核酸外切酶TREX1和TREX2会清除细胞质中游离的DNA片段从而避免细胞炎症反应的过度激活6。作者发现,在编辑过程中同时表达TREX1或TREX2可以显著降低编辑过程中线粒体DNA的插入(图二B),为解决这一编辑安全隐患提出了解决的思路。
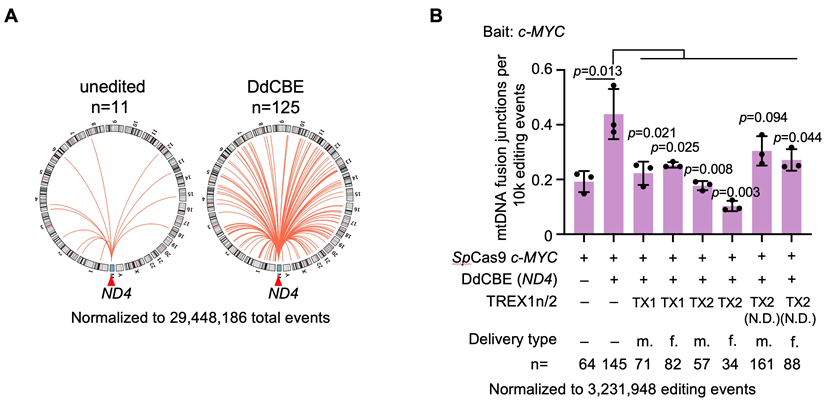
图二. 线粒体编辑器引起线粒体DNA片段整合到核基因组
该工作由生命科学学院、北大清华生命科学联合中心、北京大学成都前沿交叉生物技术院胡家志课题组和中国科学院遗传与发育生物学研究所田烨课题组合作完成。胡家志研究员为该工作的通讯作者。前沿交叉学院2023届博士毕业生吴锦淳、生命科学学院博士后刘阳(现为北京大学基础医学院PI)、PTN2021级博士研究生欧丽琼,北京大学成都前沿交叉生物技术院甘婷婷博士为论文的共同第一作者。张丁峥嵘、元绍鹏、刘心怡、刘孟竺、李佳晟、尹健行、辛昌昌亦有重要贡献。该工作得到了科技部、农业部、国家自然科学基金、蛋白质与植物基因研究国家重点实验室和细胞增殖与分化教育部重点实验室、北大-清华生命科学联合中心、生命科学学院、生命科学学院仪器平台以及凤凰中心的大力支持。
参考文献:
1. Kopinski, P.K., Singh, L.N., Zhang, S., Lott, M.T. & Wallace, D.C. Mitochondrial DNA variation and cancer. Nat Rev Cancer 21, 431-445 (2021).
2. Gray, M.W., Burger, G. & Lang, B.F. Mitochondrial evolution. Science 283, 1476-1481 (1999).
3. Wei, W. et al. Nuclear-embedded mitochondrial DNA sequences in 66,083 human genomes. Nature 611, 105-114 (2022).
4. Silva-Pinheiro, P. & Minczuk, M. The potential of mitochondrial genome engineering. Nat Rev Genet 23, 199-214 (2022).
5. Liu, Y. et al. PEM-seq comprehensively quantifies DNA repair outcomes during gene-editing and DSB repair. STAR Protocols 3, 101088 (2022).
6. Yang, Y. et al., Trex1 Exonuclease Degrades ssDNA to Prevent Chronic Checkpoint Activation and Autoimmune Disease. Cell 131, 873-886 (2007)
原文链接:https://www.nature.com/articles/s41467-024-53806-0
胡家志实验室网址:https://hulab.pku.edu.cn/